Python 解决方案:https://github.com/hardikkamboj/An-Introduction-to-Statistical-Learning
第八章 基于树的方法
-
本章介绍基于树的(tree-based)回归和分类方法,这些方法主要根据分层(stratifying)和分割(segmenting)的方式,将预测变量空间划分为一系列简单区域。对某个给定待预测的观测值,用它所属区域中的训练集的平均值或众数进行预测。
- 由于划分空间的分裂规则可以被概括为一棵树,又称作决策树(decision tree)方法。
- 基于树方法简便且易于解释,但准确率低于第六章和第七章的指导学习方法。将大量的树进行集成可以极大的提升预测准确性,但是会损失一些解释性。
8.1 决策树的基本原理
- 8.1.1 回归树(regression tree)
- 最后划分出的区域称作树的终端结点(terminal node)或树叶(leaf)。决策树通常是由上往下画的,树叶位于树的底部。
- 沿着树将预测变量空间分开的点称为内部结点(internal node),各个结点的连接部分称为分支(branch)。
- 通过特征空间分层预测
- 建立回归树的一般过程:
- 将预测变量空间分割成 J 个不重叠的区域 R1~RJ,然后对落入区域 Rj 的每个观测值进行预测(取该区域上训练集的响应值的算术平均)。
- 递归二叉分裂(recursive binary splitting):一种自上而下的、贪心的方法。从顶部开始,每个结点分割两个分支。贪心(greedy)指在建立树的每一步中,最优的分裂只取决于当前的一步,而不考虑未来。
- 树的剪枝:
- 上述方法会有良好的预测结果,但是很可能造成过拟合,原因是这样产生的树可能过于复杂。
- 裂点更少,区域数更少的树会有更小的方差和更好的解释性(以增加微小的偏差为代价)。一种策略是:仅当分裂使残差平方和的减小量达到某个阈值时,才分裂该结点。其缺点是:过于短视,因为一个结点很可能在以后的分裂中使 RSS 大度地减小。
- 更好的策略是:形成一颗较复杂的树,再通过剪枝(prune)得到子树(subtree)。但由于子树的数量可能极其庞大,对对每一棵子树都进行交叉验证会太复杂。
- 解决的方案有:代价复杂性剪枝(cost complexity pruning),也称作最弱联系剪枝(weakest link pruning)
- 8.1.2 分类树(classification tree)
- 与回归树类似,区别在于分类树用来预测定性变量。构造与回归树也类似,使用了递归二叉树分裂。但是 RSS 指标替换为分类错误率
- 8.1.3 树与线性模型的比较
- 当存在复杂的高度非线性,基于树方法将更优。
- 8.1.4 树的优缺点
- 解释性强;更接近人的决策模式;可以用图形直观表示;可以直接处理定性的预测变量而不需要创建哑变量。缺点是预测不够准确。
8.2 装袋法、随机森林和提升法
- 8.2.1 装袋法(bagging)
- 也称自助法聚集(bootstrap aggregation),类似于第五章的自助法。通过自助抽样创建初始训练集的多个副本,分别建立决策树,最后将这些树结合起来。
- 袋外误差估计
- 变量重要性的度量
- 8.2.2 随机森林(random forest)
- 通过对树作去相关处理,实现了装袋法的改进。
- 8.2.3 提升法(boosting)
- 同为装袋法的改进
8.3 实验:决策树
1
2
3
4
5
library(ISLR)
library(tree)
library(MASS)
library(randomForest)
library(gbm)
(1)构建分类树 – Carseats 数据集
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
attach(Carseats)
# 由于 Sales 为连续变量,需要记为二元变量
High <- ifelse(Sales <= 8, 'No', 'Yes')
# 加入原数据集
Carseats <- data.frame(Carseats, High)
# 分集
set.seed(1)
train <- sample(1:nrow(Carseats), 200)
Carseats.test <- Carseats[-train, ]
High.test <- High[-train]
# 使用除了 Sales 外的所有变量拟合 High 变量,tree() 的语法与 lm() 类似
tree.carseats <- tree(High~.-Sales, data=Carseats, subset=train)
tree.pred <- predict(tree.carseats, Carseats.test, type='class')
table(tree.pred, High.test)
plot(tree.carseats)
text(tree.carseats, pretty=0)
# 尝试进行减枝优化模型
set.seed(2)
# 交叉验证确定最优树
cv.caeseats <- cv.tree(tree.carseats, FUN=prune.misclass)
names(cv.carseats) #分别给出叶子数、错误率、复杂性参数值、剪枝方法
# 画出错误率 dev 对 size 和 k 的函数
par(mfrow=c(1,2))
plot(cv.carseats$size, cv.carseats$dev, type='b')
plot(cv.carseats$k, cv.carseats$dev, type='b')
# 树越大错误率越低,复杂度越高
(2)构建分类树 – Boston 数据集
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
# 创建训练集并根据训练集生成树
set.seed(3)
train <- sample(1:nrow(Boston), nrow(Boston)/2)
tree.boston <- tree(medv~., Boston, subset=train)
summary(tree.boston)
# 画出树
plot(tree.boston)
text(tree.boston, pretty=0)
# 交叉验证并生成剪枝树
cv.boston <- cv.tree(tree.boston)
plot(cv.boston$size, cv.boston$dev, type='b')
prune.boston <- prune.tree(tree.boston, best=5)
plot(prune.boston)
text(prune.boston, pretty=0)
# 用未剪枝的树对测试集进行预测
yhat <- predict(tree.boston, newdata=Boston[-train, ])
boston.test <- Boston[-train, 'medv']
abline(0, 1)
mean((yhat-boston.test)^2)
(3)装袋法与随机森林
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
# 装袋法:随机森林的一种特例
set.seed(4)
bag.boston <- randomForest(medv~., data=Boston, subset=train, mtry=13,
importance=TRUE)
# mtry=13 意味着每个结点要考虑全部的 13 个变量。
# 使用测试集进行评估:
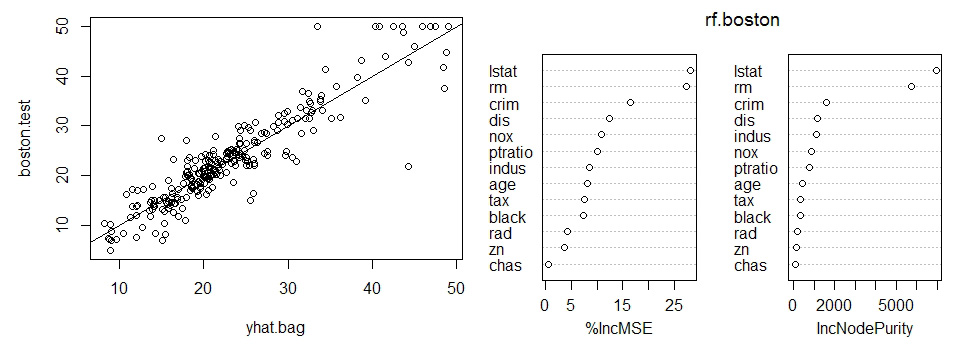
yhat.bag <- predict(bag.boston, newdata=Boston[-train, ])
plot(yhat.bag, boston.test)
abline(0, 1)
mean((yhat.bag-boston.test)^2) # 13.86875
# 使用ntree参数改变生成树的数量
bag.boston <- randomForest(medv~., data=Boston, subset=train, mtry=13,
ntree=25)
yhat.bag <- predict(bag.boston, newdata=Boston[-train, ])
mean((yhat.bag-boston.test)^2) # 14.64803
# 生成随机森林与装袋法一样,只不过 mtry 小于变量数量,默认为 p/3
set.seed(5)
rf.boston <- randomForest(medv~., data=Boston, subset=train, mtry=6,
importance=TRUE)
yhat.rf <- predict(rf.boston, newdata=Boston[-train, ])
mean((yhat.rf-boston.test)^2) # 13.72421
# 使用 importance 函数查看个变量的重要性,并绘图显示
importance(rf.boston)
varImpPlot(rf.boston)
(4)提升法
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
" 用 gbm 包对 Boston 数据集建立回归树
由于是回归问题,gbm() 函数的 distribution 参数选用'gaussian'
如果是二分类问题则选用 'bernoulli'
对象 n.tree=5000 表示提升法模型共需要 5000 棵树
选项 interaction.depth=4 限制每棵树的深度。
"
library(gbm)
set.seed(1)
boost.boston <- gbm(medv~., data=Boston[train, ],
distribution='gaussian',
n.trees=5000,
interaction.depth=4)
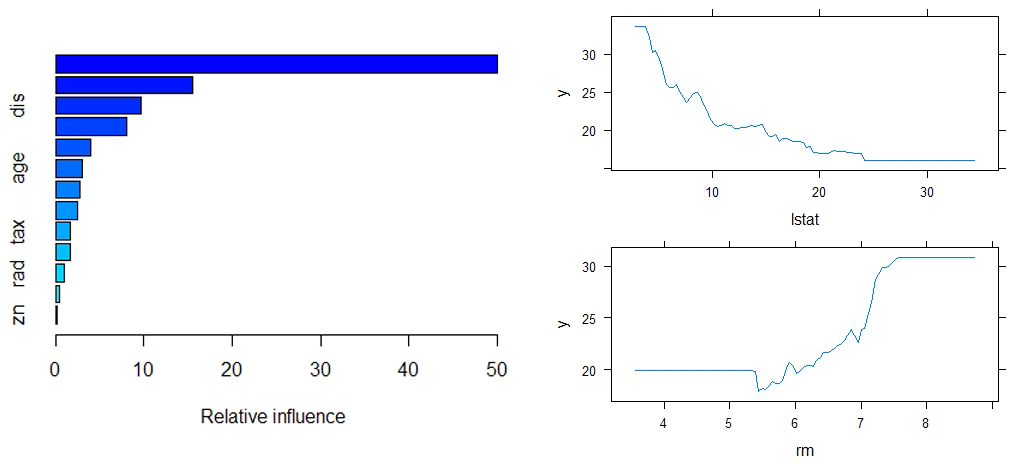
# 使用summary生成相对影响图,并输出相对影响统计数据
summary(boost.boston)
# 画出最重要的两个变量的偏相关图(类似偏导数)
par(mfrow=c(2,1))
plot(boost.boston, i='rm')
plot(boost.boston, i='lstat')
# 使用测试集进行评估
yhat.boost <- predict(boost.boston, newdata=Boston[-train, ],
n.trees=5000)
mean((yhat.boost-boston.test)^2) # 16.50313
# 结果与随机森林接近
# 使用不用的压缩参数 lambda 进行提升法,默认值是 0.001,现在取 0.2
boost.boston <- gbm(medv~., data=Boston[train, ],
distribution='gaussian',
n.trees=5000, interaction.depth=4, shrinkage=0.2,
verbose=FALSE)
# 测试均方差略低于默认压缩率
第九章 支持向量机
支持向量机(support vector machine, SVM)是一种分类方法,在许多问题有较好的效果,是适应性最广的分类器之一。可以看作一类简单直观的最大间隔分类器(maximal margin classifier)。
9.1 最大间隔分类器
- 9.1.1 超平面(hyperplane)
- 二维空间的超平面是一条直线,三维空间的超平面是一个平面。也就是说,在 p 维空间中,超平面是 p-1 维的平直的子空间。
- 一个 p 维空间的超平面的表达式:
beta0 + beta1*X1 + beta2*X2 + ... + beta_p*X_p = 0
- 使上式大于 0 的 X 落在超平面的一侧,使上式小于 0 的 X 落在另一侧
- 9.1.2 使用分割超平面分类
- 根据训练数据以某种法则找出一个超平面,即一个线性决策边界。进行预测时,测试数据被分到哪一类取决于观测点落到超平面的哪一侧。
- 9.1.3 最大间隔分类器
- 由于可以找到无数个超平面分割数据,因此需要一个选择标准。一种方法就是最大间隔分类器,也称最优分离超平面(optimal separating hyperplane)
- 具体操作:
- 首先计算训练集每个观测到一个特定的超平面的垂直距离,这些距离的最小值就是训练观测与分割超平面的距离,称作间隔(margin)
- 最大间隔超平面就是使间隔最大化的一个分割超平面。
- 距离分割超平面最近的观测点称作支持向量。
- 9.1.4 构建最大间隔分类器
- 9.1.5 线性不可分的情况
- 在许多情况下并不存在分割超平面,因此需要支持向量分类器
9.2 支持向量分类器
- 9.2.1 概述
- 即使存在分割超平面,上述分类器也经常不可取,因为对观测个体太敏感。
- 支持向量分类器,也称软间隔分类器(soft margin classifier),允许小部分观测被错误分类,而保证分类器的稳定性。
- 9.2.2 细节
- 落在间隔上或者落在错误的一侧的观测称为支持向量,因为只有这些观测会影响支持向量分类器。
- 支持向量分类器只有一部分观测(支持向量)确定,意味着对于距离超平面较远的观测来说,分类器是鲁棒的。
9.3 狭义的支持向量机 – 由线性决策边界过渡到非线性
- 9.3.1 使用非线性决策边界分类
- 实际中会遇到非线性的分类。类似回归问题中使用多项式函数的非线性扩展,在支持向量分类器中可以类似地处理非线性问题。
- 例如使用二次多项式或更高阶的多项式扩大特征空间,对于加入二次多项式,原来的 p 个特征将变成 2p 个特征,然后可以在更高维的空间中生成线性的决策边界。
- 9.3.2 支持向量机
- SVM 是支持向量分类器的一种扩展,使用了核函数(kernel)来扩大特征空间。
- 核函数的使用
- 9.3.3 心脏数据集的应用
9.4 多分类的SVM
- 扩展到 K 类主要有两种方法:一类对一类(One versus one),一类对余类。
9.5 与逻辑斯谛回归的关系
9.6 实验:支持向量机
1
2
3
# e1071 包或者 LiblineaR 包都可以实现支持向量分类器和 SVM
library(ISLR)
library(e1071)
(1)支持向量分类器
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
# 首先生成属于两个类别的观测数据
set.seed(1)
x <- matrix(rnorm(20*2), ncol=2)
y <- c(rep(-1, 10), rep(1, 10))
x[y==1, ] <- x[y==1, ] + 1
# 检查是线性可分,结果非线性可分
plot(x, col=y+3)
# 构建数据集,将响应变量转换成因子变量
data <- data.frame(x=x, y=as.factor(y))
# 拟合支持向量分类器
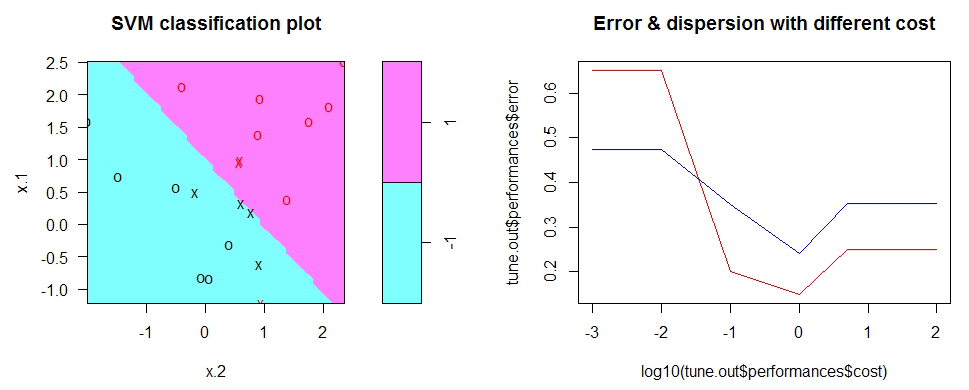
svm.fit <- svm(y~., data=data, kernel='linear', cost=10, scale=FALSE)
# 可视化分类器
plot(svm.fit, data)
# 查看支持变量
svm.fit$index
# 使用更小的 cost 参数,可以得到更宽的间隔,更多的支持变量
# 使用 tune 函数进行交叉验证调参
set.seed(2)
tune.out <- tune(svm, y~., data=data, kernel='linear',
ranges = list(cost=c(0.001, 0.01, 0.1, 1, 5, 10, 100))
)
plot(log10(tune.out$performances$cost), tune.out$performances$error, type='l', col='red')
points(log10(tune.out$performances$cost), tune.out$performances$dispersion, type='l', col='blue')
title(main='Error & dispersion with different cost')
====================================================================
> summary(tune.out$best.model)
Call:
best.tune(method = svm, train.x = y ~ ., data = data,
ranges = list(cost = c(0.001, 0.01, 0.1, 1, 5, 10, 100)),
kernel = "linear")
Parameters:
SVM-Type: C-classification
SVM-Kernel: linear
cost: 1
Number of Support Vectors: 11
( 6 5 )
Number of Classes: 2
Levels: -1 1
====================================================================
(2)支持向量机
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
" 使用带核函数的支持向量机:
kernel='polynomial -- 拟合多项式核函数的 SVM,调节 degree 参数;
kernel='radial' -- 拟合径向基核函数的 SVM,调节 gamma 参数。
"
set.seed(3)
# 构建具有非线性决策边界的二维特征数据
x <- matrix(rnorm(200*2), ncol=2)
x[1:100, ] <- x[1:100, ] + 2
x[101:150, ] <- x[101:150, ] - 2
y <- c(rep(1, 150), rep(2, 50))
data <- data.frame(x=x, y=as.factor(y))
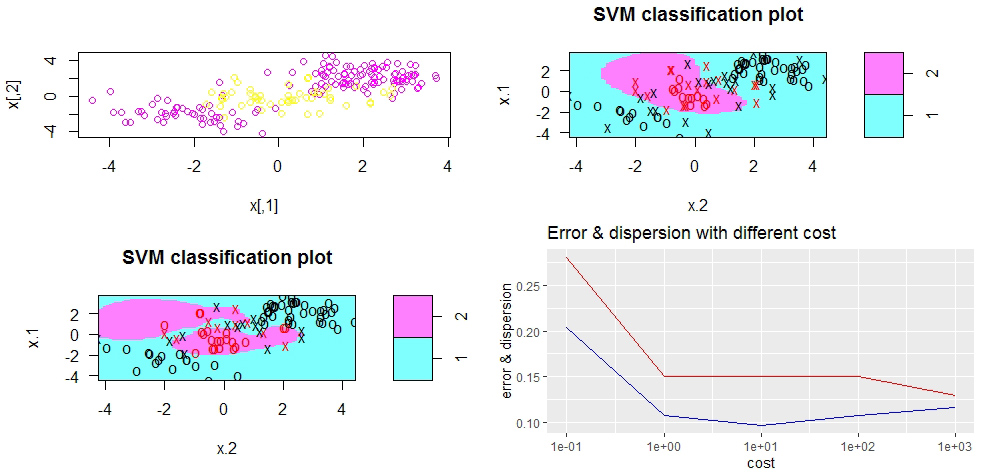
plot(x, col=y+5)
# 分集并拟合,使用径向基核函数,取 gamma=1
train <- sample(200, 100)
svm.fit <- svm(y~., data=data[train, ], kernel='radial', gamma=1, cost=1)
plot(svm.fit, data[train, ])
# 使用更大的 cost 减少误分观测,代价是边界不规则,可能会过拟合
svm.fit <- svm(y~., data=data[train, ], kernel='radial', gamma=1, cost=1e5)
plot(svm.fit, data[train, ])
# 使用 tune 进行交叉验证确定最佳的 gamma 和 cost 值
set.seed(4)
tune.out <- tune(svm, y~., data=data[train, ], kernel='radial',
ranges=list(cost=c(0.1, 1, 10, 100, 1e3)),
gamma=c(0.5, 1, 2, 3, 4))
library(tidyverse)
tune.out$performances %>% ggplot(aes(x=cost)) +
geom_line(aes(y=error), color='red') +
geom_line(aes(y=dispersion), color='blue') +
scale_x_log10() +
labs(y = 'error & dispersion',
title = 'Error & dispersion with different cost')
===========================================
> summary(tune.out)
Parameter tuning of ‘svm’:
- sampling method: 10-fold cross validation
- best parameters:
cost
1000
- best performance: 0.13
- Detailed performance results:
cost error dispersion
1 1e-01 0.28 0.20439613
2 1e+00 0.15 0.10801234
3 1e+01 0.15 0.09718253
4 1e+02 0.15 0.10801234
5 1e+03 0.13 0.11595018
===========================================
# 使用最佳参数在测试集上进行评估
table(tune = dat[-train, 'y'],
pred = predict(tune.out$best.model, newx=data[-train, ]))
(3) ROC 曲线( Receiver Operating Characteristic)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
library(ROCR)
# 定义一个函数,在给定的包含每个观测值的 pred 和真实值下,画出ROC曲线
rocplot <- function(pred, truth, ...){
predob <- prediction(pred, truth)
perform <- performance(predob, 'tpr', 'fpr')
plot(perform, ...)
}
# 拟合模型
svmfit.opt <- svm(y~., data=dat[train, ],
kernel='radial', gamma=2, cost=1, decision.values=TRUE)
fitted <- attributes(predict(svmfit.opt, dat[train, ],
decision.values=TRUE))$decision.values
# 绘制ROC曲线
rocplot(fitted, dat[train, 'y'], main='Training Data')
(4)多分类 SVM
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
" 使用khan数据集,数据集由 2308 个基因的表达测定组成;
训练集和测试集分别由 63 和 20 个观测组成;"
names(Khan)
dim(Khan$xtrain)
dim(Khan$xtest)
table(Khan$ytrain)
table(Khan$ytest)
# 使用支持向量机预测癌症,由于特征数特别多,使用线性核函数
data <- data.frame(x=Khan$xtrain, y=as.factor(Khan$ytrain))
out <- svm(y~., data=data, kernel='linear', cost=10)
summary(out)
table(out$fitted, data$y)
# 可以看到训练集误差为 0,因为特征空间维度高,产生过拟合
# 测试集误差
data.test <- data.frame(x=Khan$xtest, y=as.factor(Khan$ytest))
pred.test <- predict(out, newdata=data.test)
table(pred.test, data.test$y)
第十章 无监督学习(unsupervised learning)
无监督学习是一系列统计工具,研究只有特征数据而没有响应变量的情况。其目标并非预测,而是寻找特征空间的有价值的模式。常见两种无监督学习:主成分分析(PCA)、聚类分析(clustering)。
10.1 无监督学习的挑战
相比于前面介绍的有监督学习,无监督学习更具有挑战性。训练更倾向于主观性,不设定明确的分析目标。评价一个无监督学习的结果非常困难。
10.2 主成分分析
第六章研究主成分回归(PCR)时,解释了主成分方向:特征空间中原始数据高度变异(highly variable)的方向。获取主成分使用了 PCA 方法。
-
10.2.1 什么是主成分
- 如果要将 p 个特征画出相关矩阵散点图,将有 p(p-1)/2 个散点图。而且每个图只包含数据集很小一部分信息,没有多大价值。使用 PCA 就可以得到一个二维表示来获得数据集的大部分信息。
- 基本思想:n 个观测虽然都在 p 维空间中,但不是所有的维度同样地有价值。PCA 寻找尽可能少的有意义的维度(离散度越高越有价值)。通过PCA找到的每个维度都是原始的 p 个特征的线性组合。
- 第一主成分是变量的标准化线性组合中方差最大的组合:
Z1 = phi_11*X_1 + phi_21*X_2 + ... + phi_p1*X_p- phi是希腊字母,代表载荷(loading)。标准化(normalized)是指为了防止载荷大小影响方差大小,限定载荷的平方和为 1。寻找第一主成分可以理解为在上述约束下最大化载荷平方和。
-
10.2.2 主成分的另一种解释
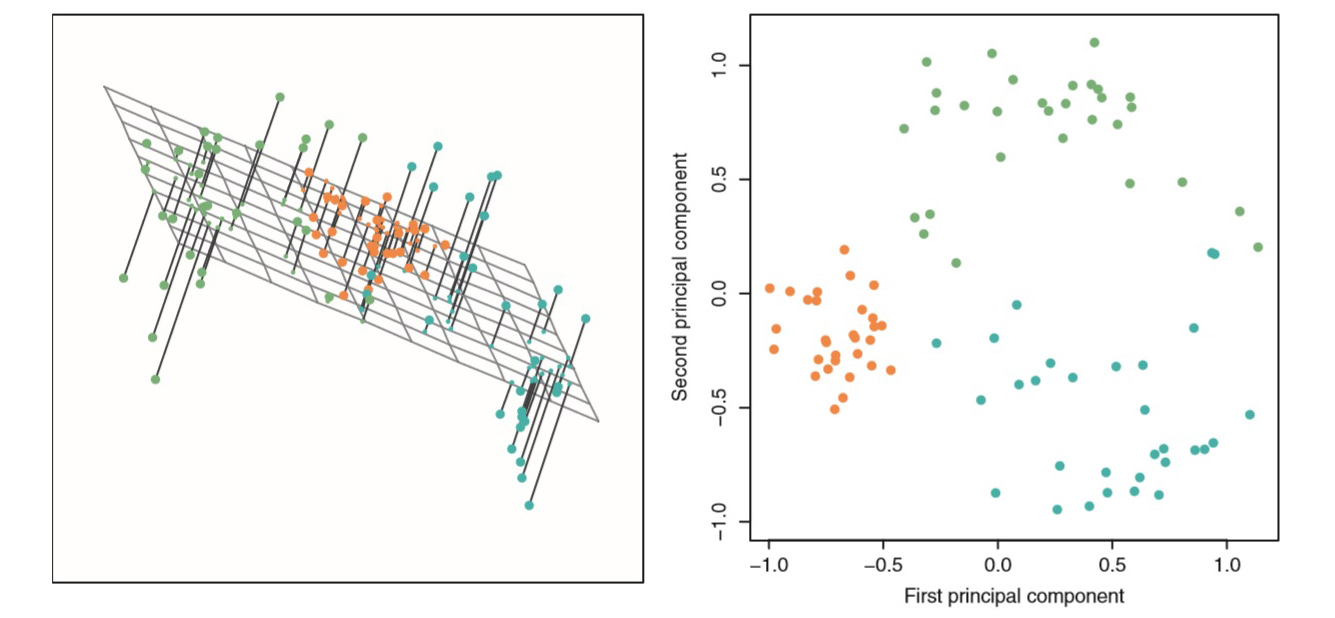
- 上图显示了一个三维数据集的前两个主成分载荷向量。这两个主成分张成一个平面,这个平面在空间中的方向是观测数据方差达到最大的方向。三维数据从主成分载荷方向投影到一个二维平面,得到一张二维图,即主成分得分向量。
- 对此得到主成分分析的另一种解释:主成分提供了一个与观测数据最接近的低维线性空间(我理解为面积最小的投影)。
-
10.2.3 关于 PCA 的其他方面
- 变量的标准化:在进行 PCA 之前,变量应该中心化使均值为零,还需要进行变量的标准化,某则影响结果。这是 PCA 与其他方法的重要区别。例如线性回归中,变量是否标准化对结果没有影响。
- 主成分的唯一性:除了计算出来的符号正负不同(向量正方向不同),每个主成分的载荷向量是唯一的(即使用不同的软件包计算)。
- 方差的解释比例(proportion of variance explained, PVE):表示每个主成分解释了多少比例(百分比)的方差。计算公式略。
- 决定主成分的数量:n*p 维的数据矩阵 X 有 min(n-1, p) 个不同的主成分,但是一般不会全部用上。用多少比较好,没有一个最佳答案。
-
10.2.4 主成分的其他用途
10.3 聚类分析方法
- clustering 是在一个数据集中寻找子群或类的方法,应用广泛。基本原则是,是每个类内的观测彼此尽量相似,不同类的差异尽量大。
- 聚类和 PCA 的目标都是用少量的概括性信息简化数据,但是机制不同:PCA 寻找观测的一个低维表示来解释大部分方差,聚类从观测中寻找同质子类。
-
主要聚类方法:K 均值聚类(K-means clustering)和系统聚类(hierarchical clustering);前者预先规定类的数量,后者反之。
-
10.3.1 K 均值聚类
- K-means 算法:
- 1.确定 K 值,为每个观测随机初始化 1~K 的数值
- 2.重复以下直到收敛:
- a.分别计算 K 个类的中心,即该类中 p 维观测向量的均值向量
- b.将每个观测分配到距离最近的类中心所属的类(欧氏距离的“最近”)
- 由于 K-means 算法找到的是局部最优解,算法收敛是未必是总体最优解,还跟初始化有关。因此需要进行随机初始化,多次运行再对比找到最优解。
- K-means 算法:
-
10.3.2 系统聚类法
- 与 K-means 聚类对比,系统聚类法不需要预设类数 K,另外一个优点是可以输出一个好看的有关各观测的树型表示,称作谱系图(dendrogram)
- 一种常见的系统聚类方法:自下而上(bottom-up)的方法,也称凝聚法(agglomerative)。其谱系图由叶子开始聚集到树干,形成一棵倒过来的树。
- 解释谱系图:通过谱系图直观解释系统聚类法,略。
- 算法实现:
-
- 计算 n 个观测中所有 n(n-1)/2 对观测数据之间的相异度(如欧氏距离),将每个观测看作一类。
-
- 令 i = n, n-1, n-2, … ,2:
- a.在 i 个类中,比较任意两个类的相异度,找到相异度最小的一对,并结合。用两个类之间的相异度表示两个类在谱系图中交汇的高度
- b.计算剩余的 i-1 个类中,每两个类的相异度
- 令 i = n, n-1, n-2, … ,2:
- LOOP
-
- 四种常见的距离形式:最长距离法、最短距离法、类平均法、重心法。
- 相异度指标的选择:一般都是欧氏距离
- 10.3.3 聚类分析的实践问题
- 小策略撬动大理论:涉及很多方法或指标选择的问题
- 验证聚类结果的问题:得到的是代表数据共性的子类,还是仅仅将噪声聚到了一起?可以通过 p 值进行显著性检验,但是没有公认的好方法。
- 聚类分析的其他考虑:
- 解释聚类分析结果的一个折中方法:建议对数据集的子集进行聚类分析,这样可以对所得到的类的稳定性有一个整体感知。
10.4 实验
(1)主成分分析
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
"使用 USArrests 数据集进行 PCA, 数据集包含50个州的观测,4个变量"
library(ISLR)
states <- row.names(USArrests)
variables <- names(USArrests)
# 查看变量的均值和方差,发现相差较大
apply(USArrests, 2, mean)
apply(USArrests, 2, var)
# 因此使用带有标准化的主成分分析
pr.out <- prcomp(USArrests, scale=TRUE)
pr.out$rotation # 主成分载荷向量
pr.out$x # 主成分得分向量
pr.out$sdev # 主成分标准差
# 绘出前两个主成分的双标图
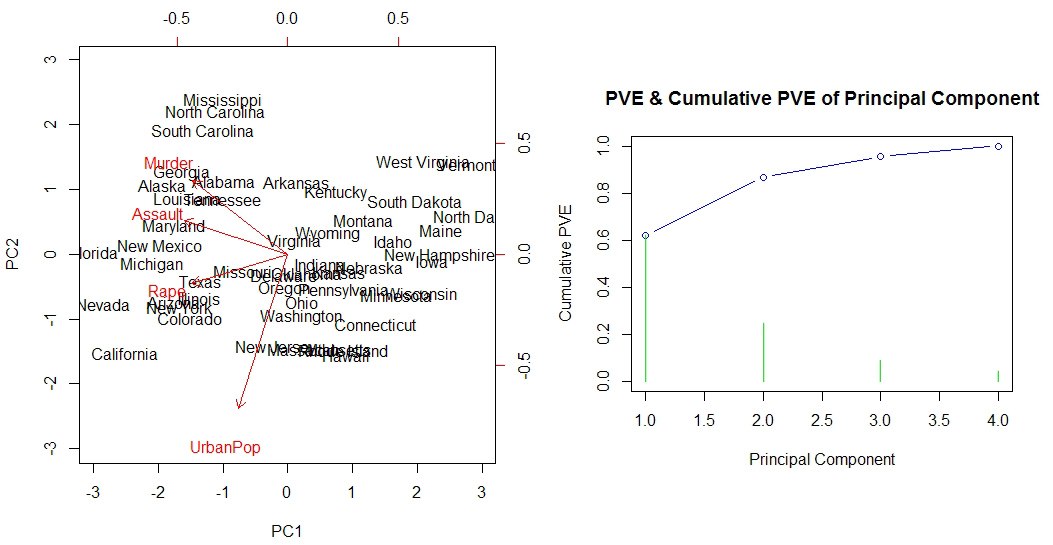
biplot(pr.out, scale=0)
# 计算每个主成分的方差解释比
pve <- pr.out$sdev^2 / sum(pr.out$sdev^2)
# 绘制每个主成分的 PVE 和累积 PVE
plot(cumsum(pve),
xlab='Principal Component', ylab='Cumulative PVE',
ylim=c(0,1), type='b', col='blue')
points(pve, ylim=c(0,1), type='h', col='green')
title(main='PVE & Cumulative PVE of Principal Component')
(2)聚类分析
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
"------------------------------- K Means -------------------------------"
# 创建数据
set.seed(1)
x <- matrix(rnorm(50*2), ncol=2)
x[1:25, 1] <- x[1:25, 1] + 3
x[1:25, 2] <- x[1:25, 2] - 3
# 使用较大的 nstart,可以模拟较多的随机初始化,并选取最佳结果
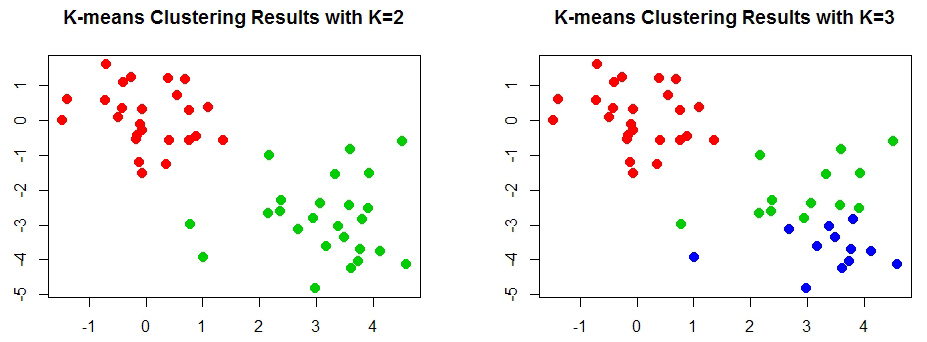
km.out <- kmeans(x, 2, nstart=20)
# 可视化模型结果
plot(x, col=(km.out$cluster+1),
main='K-means Clustering Results with K=2',
xlab='', ylab='', pch=20, cex=2)
# 注意到数据是二维的,容易可视化。对于多位数据,可以绘制前两个主成分的得分向量
# 对数据进行K=3的K均值聚类
set.seed(2)
km.out<-kmeans(x, 3, nstart=20)
plot(x, col=(km.out$cluster+1),
main='K-means Clustering Results with K=3',
xlab='', ylab='', pch=20, cex=2)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
"------------------------------ 系统聚类法 ------------------------------"
# 使用 hclust 函数进行分层聚类,使用不同的距离方式,以欧式距离作为相异度
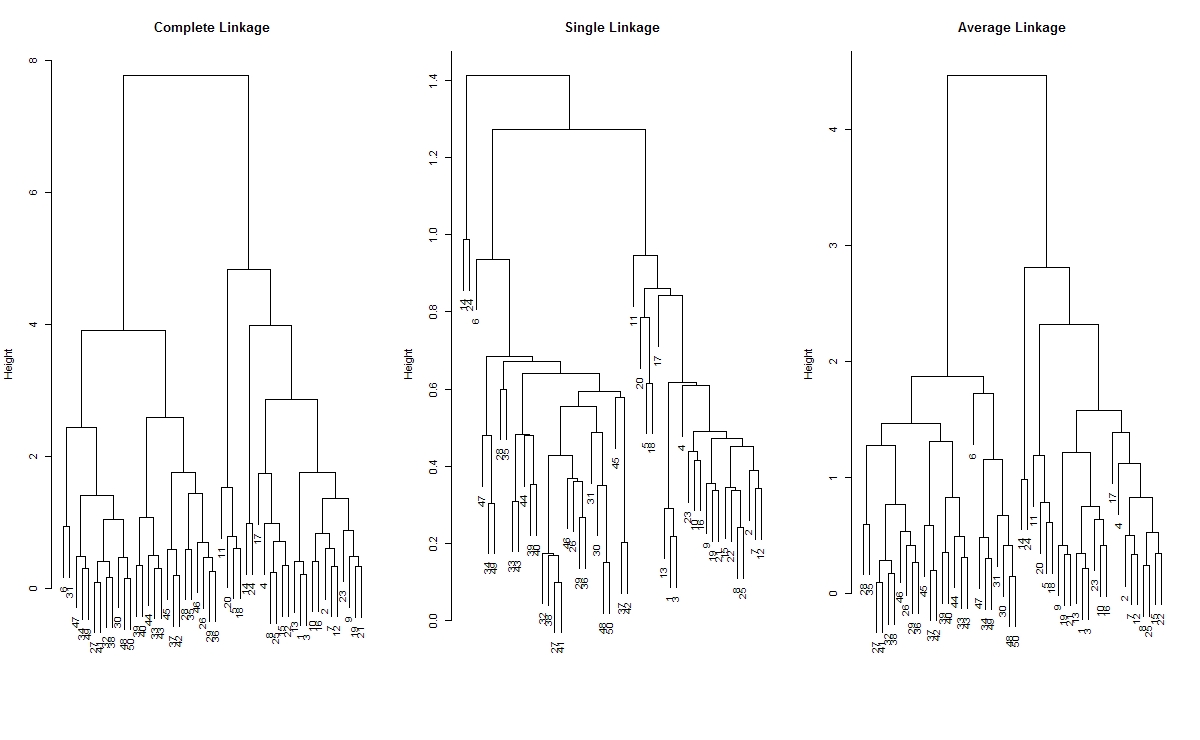
hc.complete <- hclust(dist(x), method='complete') # 最长距离法
hc.single <- hclust(dist(x), method='single') # 最短距离法
hc.average <- hclust(dist(x), method='average') # 类平均法
# 绘制谱系图,尽头的每个数字代表一个观测
par(mfrow=c(1,3))
plot(hc.complete, main='Complete Linkage', xlab='', sub='', cex=0.9)
plot(hc.single, main='Single Linkage', xlab='', sub='', cex=0.9)
plot(hc.average, main='Average Linkage', xlab='', sub='', cex=0.9)
# 使用 cutree 函数根据谱系图的切割获取各个观测的类标签
========================================================================
> cutree(hc.complete, 2)
[1] 1 1 1 1 1 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2
[35] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
========================================================================
1
2
3
4
5
6
7
8
9
10
11
12
# 对标准化的数据进行系统聚类
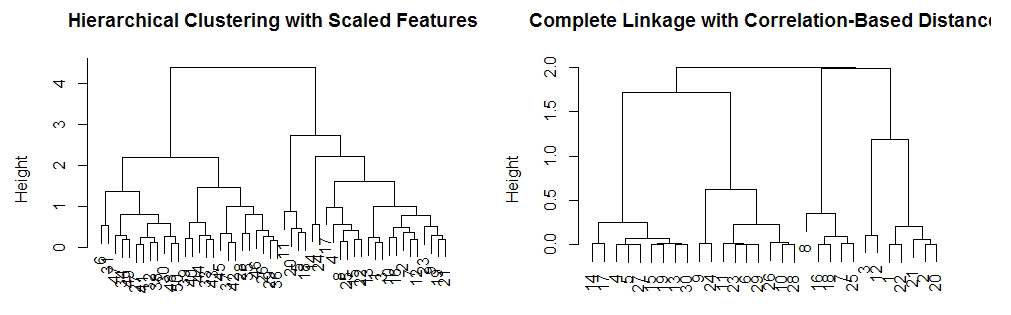
x_sc <- scale(x)
plot(hclust(dist(xsc), method='complete'),
main='Hierarchical Clustering with Scaled Features',
xlab='', sub='')
# 修改欧氏距离为基于相关性距离,使用 as.dist() 函数
x <- matrix(rnorm(30*3), ncol=3)
dd <- as.dist(1-cor(t(x)))
plot(hclust(dd, method='complete'),
main = 'Complete Linkage with Correlation-Based Distance',
xlab='', sub='')
(3)以 NCI60 数据集为例
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
# PCA 和系统聚类法常用于基因数据的分析
# NCI60 数据集由 64 个细胞的共 6830 个基因表达数据组成
nci.labs <- NCI60$labs # 癌细胞类型
table(nci.labs)
nci.data <- NCI60$data # 基因数据
dim(nci.data)
"--------------------- 使用主成分分析查找癌症相关细胞 ---------------------"
# 数据标准化的 PCA
pr.out <- prcomp(nci.data, scale=TRUE)
# 定义函数给每个观测对应的癌症类型分配不同颜色
Cols <- function(vec){
cols <- rainbow(length(unique(vec)))
return (cols[as.numeric(as.factor(vec))])
}
# 对前几个主成分进行可视化,绘制得分向量图
par(mfrow=c(1,2))
plot(pr.out$x[,1:2], col=Cols(nci.labs), pch=19, xlab='Z1', ylab='Z2')
plot(pr.out$x[,c(1,3)], col=Cols(nci.labs), pch=19, xlab='Z1', ylab='Z3')
# 可以看到对于同类癌症的细胞系的前几个主成分得分向量比较接近
# 绘制前几个主成分解释的方差
plot(pr.out)
# PVE 和累计 PVE
pve <- pr.out$sdev^2 / sum(pr.out$sdev^2)
plot(cumsum(pve), xlab='', ylab='', ylim=c(0,1), type='l', col='blue')
points(pve, xlab='', ylab='', ylim=c(0,1), type='h', col='green')
title(main='PVE & Cumsum-PVE')
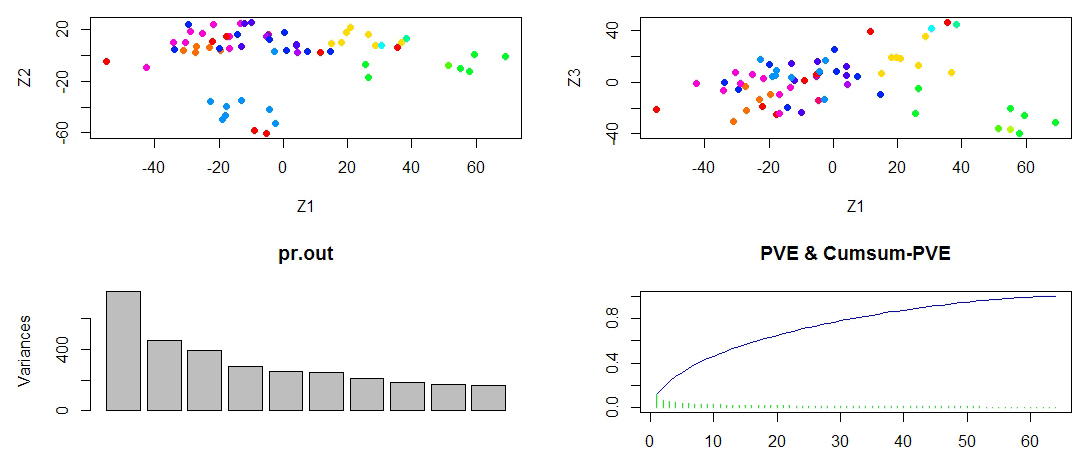
1
2
3
4
5
6
7
8
9
10
11
12
13
"---------------------- 对癌症的基因表现进行聚类分析 ----------------------"
sc.data <- scale(nci.data)
# 进行系统聚类并绘制谱系图
par(mfcol=c(1,3))
data.dist <- dist(sc.data)
plot(hclust(data.dist), labels=nci.labs,
main='Complete Linkage', xlab='', sub='', ylab='')
plot(hclust(data.dist, method='average'), labels=nci.labs,
main='Average Linkage', xlab='', sub='', ylab='')
plot(hclust(data.dist, method='single'), labels=nci.labs,
main='Single Linkage', xlab='', sub='', ylab='')
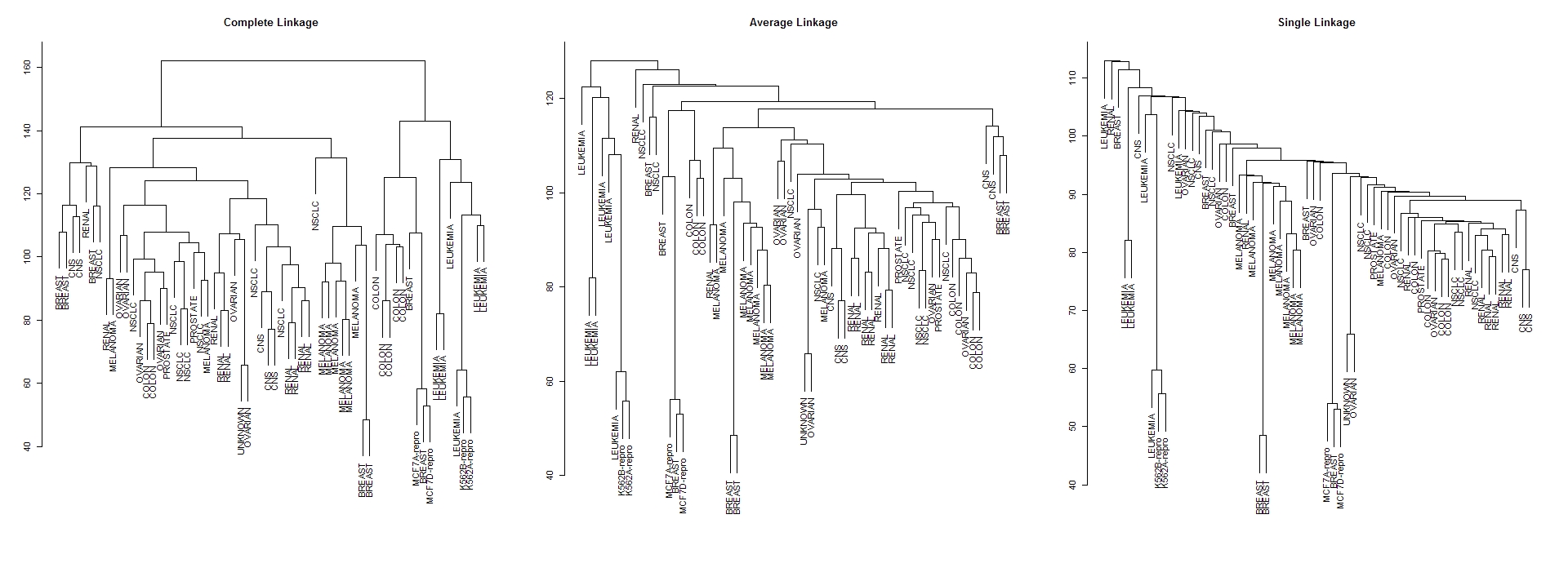
1
2
3
4
5
6
7
8
9
# 在谱系图上某个高度切割可以产生指定类数的聚类,例如 4 类
hc.out <- hclust(dist(sc.data), method='complete')
hc.clusters <- cutree(hc.out, 4)
table(hc.clusters, nci.labs)
# 绘制这四个类的谱系图切割位置
par(mfrow=c(1,1))
plot(hc.out, labels=nci.labs, xlab='', sub='', ylab='')
abline(h=139, col='red')
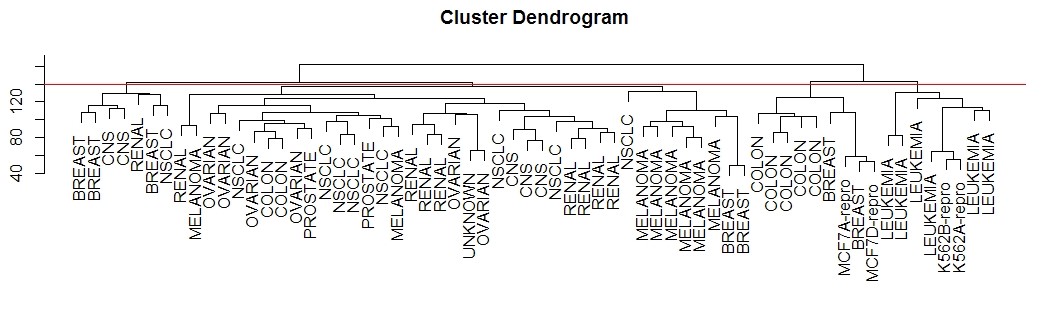
1
2
3
4
5
6
7
# 只对前几个主成分得分向量进行系统聚类,而不是全部变量
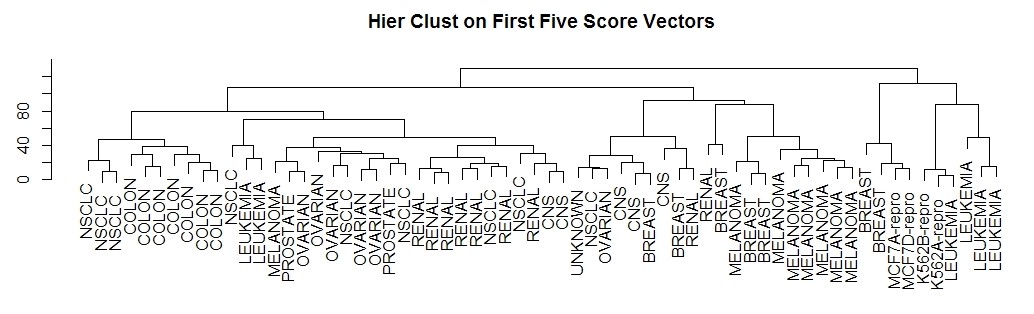
hc.out <- hclust(dist(pr.out$x[, 1:5]))
plot(hc.out, labels=nci.labs,
main='Hier Clust on First Five Score Vectors',
xlab='', sub='', ylab='')
table(cutree(hc.out, 4), nci.labs)
# 可将主成分获取的操作,看成是一种去噪处理。
END